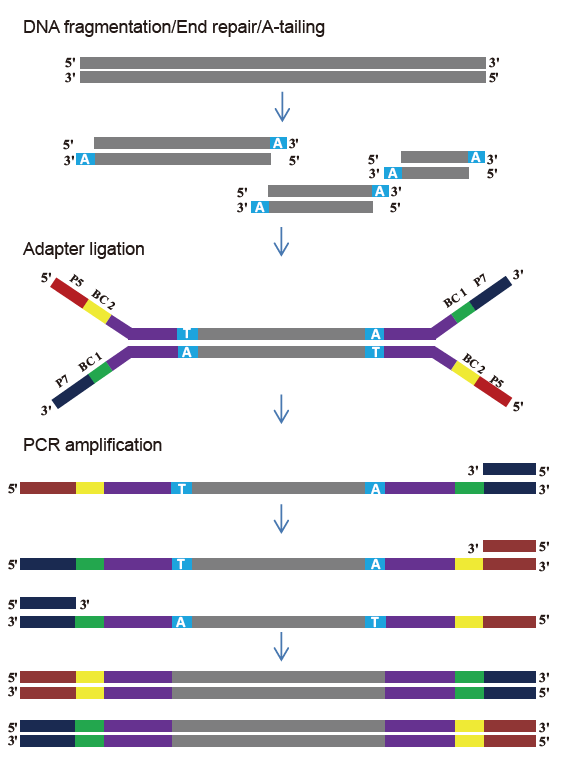
Sada knihovny TIANSeq DirectFast (osvětlení)
Funkce
■ Dobrá uniformita sekvenování: Žádná předpojatost bází při procesu fragmentace DNA a procesu amplifikace PCR.
■ Vysoká účinnost převodu knihovny: konstrukci vysoce účinné knihovny lze zajistit pro 1 ng vzorků DNA.
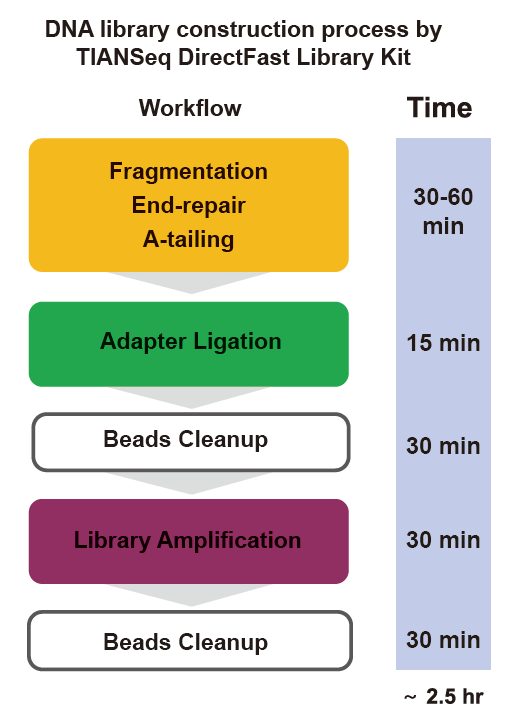
■ Rychlý provoz: Celý proces výstavby knihovny vyžaduje pouze 2,5 hodiny.
■ Cenově výhodné: Nejsou potřeba žádné speciální nástroje a vybavení。
Specifikace
Typ: Příprava knihovny DNA pro vysoce výkonnou sekvenační platformu Illumina
Vzorek: Genomická DNA nebo DNA velkého fragmentu
Cílová: Dvouvláknová DNA
Spuštění ukázkového vstupu: 1 ng- 1 μg
Doba provozu: 2,5 hodiny
Následné aplikace: Sekvenování na platformě Illumina
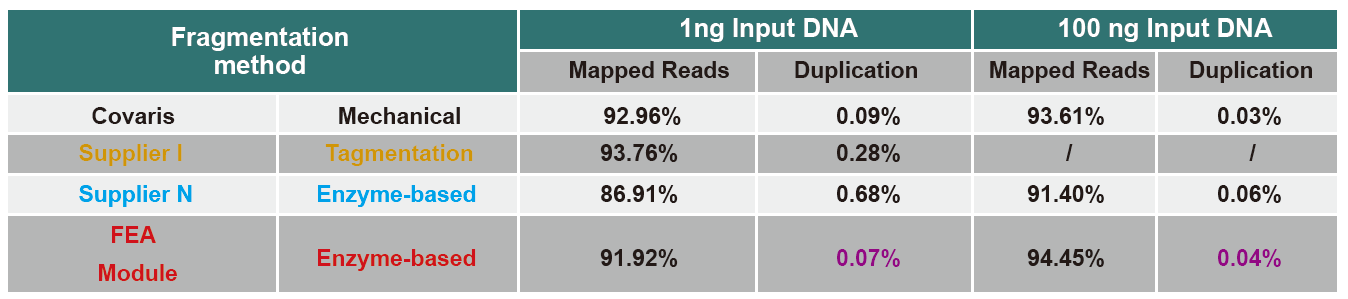
Všechny produkty lze přizpůsobit pro ODM/OEM. Pro detaily,klikněte prosím na přizpůsobenou službu (ODM/OEM)
Flexibilní vstup vzorku a fragmentovaná velikost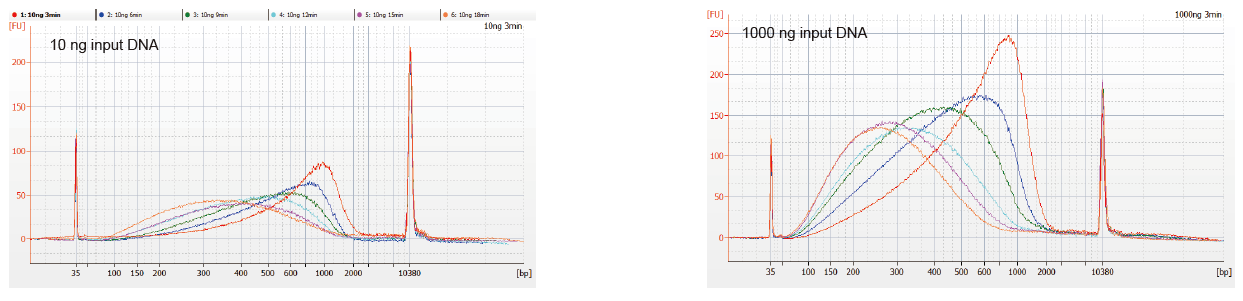 |
Obrázek 1. Profily fragmentace DNA různé reakční doby. 10 ng a 1 000 ng DNA bylo fragmentováno pomocí TIANSeq DirectFast DNA Library Kit. Reakční produkty ošetřené s různou reakční dobou byly čištěny magnetickými kuličkami 1,8 x Ampure XP a analyzovány pomocí Angilent 2100. |
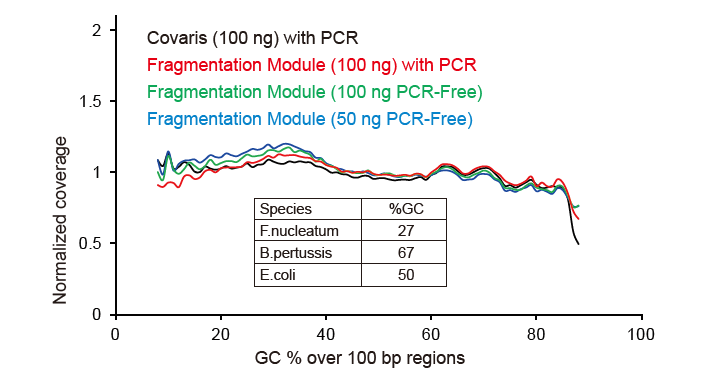
Pokrytí sekvenování podobné Covaris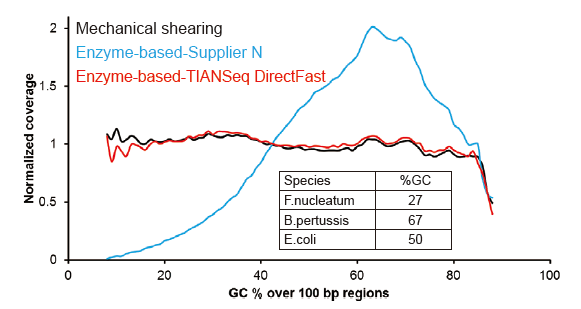 |
Obrázek 2. Srovnání pokrytí genomu různými metodami přípravy knihovny. Tři bakteriální genomové DNA s různým obsahem GC jsou smíšeny ekvimolárně a byl porovnán výsledek pokrytí sekvenování genomu 100 ng knihoven smíšených DNA pomocí těchto metod. Výsledky ukazují, že souprava TIANSeq DirectFast Library Kit má stejný účinek na fragmentaci DNA jako mechanické stříhání a neexistuje zde žádná předpětí báze pro fragmentaci. |
Žádné systematické zkreslení již od 1 ng vstupní DNA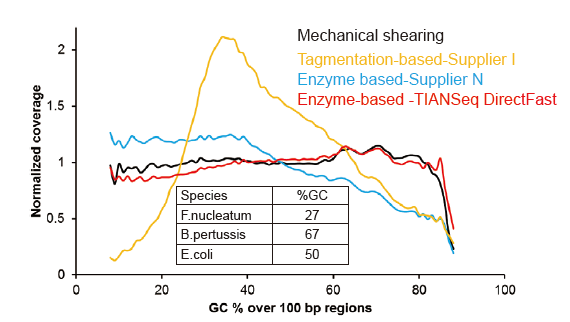 |
Obrázek 3. Porovnání pokrytí genomu různými metodami přípravy knihovny. Tři bakteriální genomové DNA s různým obsahem GC jsou smíchány ekvimolárně a byl porovnán výsledek pokrytí sekvenování genomu 1 ng knihoven smíšených DNA pomocí těchto metod. Výsledky ukazují, že souprava TIANSeq DirectFast Library Kit má konzistentní fragmentační účinek s mechanickým střihem i pro vstup DNA od 1 ng a nedochází k žádnému zkreslení báze. |
| Možnost pracovního postupu bez PCR
|
Obrázek 4. Různé vstupy genomové DNA byly použity ke konstrukci knihovny pomocí PCR nebo konstrukce knihovny bez PCR a výsledky pokrytí genomu byly porovnány. Výsledky ukazují, že díky operaci s jednou zkumavkou a efektivním krokům při konstrukci knihovny si knihovna DNA zkonstruovaná pomocí sady TIANSeq DirectFast Library Kit zachovává vysokou konzistenci s mechanickým střihem v distribuci pokrytí sekvence fragmentů pro oba pracovní postupy obohacování PCR bez PCR. |
Statistiky účinnosti a výtěžnosti výstavby knihovny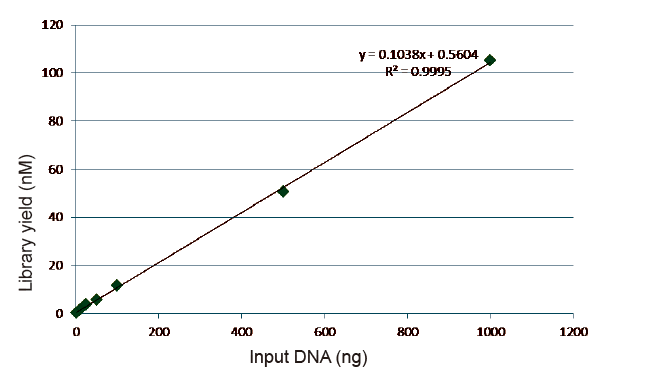 |
Obrázek 5. Výsledky kvantitativní analýzy DNA knihovny získané qPCR po konstrukci knihovny metodou bez PCR pro vzorky s různým počátečním množstvím (1, 10, 25, 50, 100, 500, 1 000 ng). Lineární regresní analýza ukazuje, že výtěžek knihovny má dobrý lineární vztah v širokém rozsahu vstupního vzorku. U vstupu DNA již od 1 ng se účinnost konstrukce knihovny nesnižuje. |
Porovnání sekvenčních dat různých produktů
V současné době je technologie sekvenování s vysokou propustností založena hlavně na technologii sekvenování nové generace. Protože je délka čtení technologie sekvenování příští generace omezená, musíme sekvenci celé délky rozdělit na malé fragmentové knihovny. Podle potřeb různých sekvenčních experimentů obvykle volíme sekvencování s jedním koncem nebo se sekvenováním s dvěma konci. V současné době jsou fragmenty DNA sekvenční knihovny příští generace obecně distribuovány v rozmezí 200 až 800 bp.

a) DNA má špatnou kvalitu a obsahuje inhibitory. Abyste zabránili inhibici aktivity enzymu, používejte vysoce kvalitní vzorky DNA.
b) Množství vzorku DNA je nedostatečné při použití metody bez PCR ke konstrukci knihovny DNA. Když vstup fragmentované DNA překročí 50 ng, lze během procesu výstavby knihovny selektivně provádět pracovní postup bez PCR. Pokud je počet kopií knihovny příliš nízký na to, aby mohl být přímo sekvenován, lze knihovnu DNA po ligaci adaptéru amplifikovat pomocí PCR.
c) Kontaminace RNA vede k nepřesné počáteční kvantifikaci DNA V procesu čištění genomové DNA může existovat kontaminace RNA, což může vést k nepřesné kvantifikaci DNA a nedostatečnému zatížení DNA během výstavby knihovny. RNA lze odstranit ošetřením RNázou.
A-1
a) Objevují se malé fragmenty (60 bp-120 bp) Malé fragmenty jsou obvykle fragmenty adaptérů nebo dimery tvořené adaptéry. Čištění pomocí magnetických kuliček Agencourt AMPure XP může tyto fragmenty adaptéru účinně odstranit a zajistit kvalitu sekvenování.
b) Velké fragmenty se objeví v knihovně po PCR amplifikaci. Velikost fragmentu DNA knihovny se po ligaci adaptéru zvýší o 120 bp. Pokud se fragment DNA po ligaci adaptéru zvýší o více než 120 bp, může to být způsobeno abnormální fragmentovou amplifikací nadměrné amplifikace PCR. Snížení počtu cyklů PCR může situaci zabránit.
c) Abnormální velikost fragmentů DNA knihovny po ligaci adaptéru Délka adaptéru v této sadě je 60 bp. Když jsou dva konce fragmentu ligovány k adaptérům, délka se zvýší pouze o 120 bp. Pokud používáte jiný adaptér, než jaký poskytuje tato sada, obraťte se na dodavatele a poskytněte mu relevantní informace, jako je délka adaptéru. Zajistěte, aby pracovní postup a operace experimentu postupovaly podle kroků popsaných v příručce.
d) Abnormální velikost fragmentu DNA před ligací adaptéru Důvod tohoto problému může být způsoben špatnými reakčními podmínkami během fragmentace DNA. Pro různé vstupy DNA by měly být použity různé reakční doby. Pokud je vstup DNA větší než 10 ng, doporučujeme jako počáteční čas optimalizace zvolit reakční dobu 12 minut a velikost fragmentu produkovaná v této době se pohybuje hlavně v rozmezí 300 až 500 bp. Uživatelé mohou zvýšit nebo snížit délku fragmentů DNA po dobu 2-4 minut podle svých vlastních požadavků, aby optimalizovali fragmenty DNA s požadovanou velikostí.
A-2
a) Fragmentační čas není optimalizován Pokud je fragmentovaná DNA příliš malá nebo příliš velká, přečtěte si pokyny pro výběr doby fragmentace uvedené v pokynech ke stanovení reakční doby a použijte tento časový bod jako kontrolu, navíc nastavte reakční systém prodlouží nebo zkrátí o 3 minuty, aby provedl přesnější úpravu doby fragmentace.
A-3
Abnormální distribuce velikosti DNA po fragmentační léčbě
a) Nesprávná metoda rozmrazování fragmentačního činidla nebo činidlo není po rozmrazení zcela smícháno. Rozmíchejte reagent 5 × Fragmentation Enzyme Mix na ledu. Po rozmrazení rovnoměrně promíchejte reagencii jemným pohybem dna zkumavky. Reagencii nevortexujte!
b) Vstupní vzorek DNA obsahuje EDTA nebo jiné znečišťující látky. Úspěch iontů solí a chelatačních činidel v kroku čištění DNA je zvláště důležitý pro úspěch experimentu. Pokud je DNA rozpuštěna v 1 × TE, proveďte fragmentaci metodou uvedenou v pokynu. Pokud je koncentrace EDTA v roztoku nejistá, doporučuje se DNA vyčistit a rozpustit v deionizované vodě pro následnou reakci.
c) Nepřesná počáteční kvantifikace DNA Velikost fragmentované DNA úzce souvisí s množstvím vstupu DNA. Před fragmentačním zpracováním je přesná kvantifikace DNA pomocí Qubit, Picogreen a dalších metod nezbytná pro stanovení přesného množství DNA v reakčním systému.
d) Příprava reakčního systému nedodržuje pokyny Příprava fragmentovaného reakčního systému musí být prováděna na ledu přísně podle pokynů. Aby byl zajištěn nejlepší účinek, měly by být všechny reakční složky umístěny na led a příprava reakčního systému by měla být provedena po úplném ochlazení. Po dokončení přípravy šleháním nebo pipetováním důkladně promíchejte. Nevírejte!
1. Nesprávná metoda míchání (vortex, násilná oscilace atd.) Způsobí abnormální distribuci fragmentů knihovny (jak ukazuje následující obrázek), což ovlivní kvalitu knihovny. Proto při přípravě reakčního roztoku Fragmentation Mix jemně pipetujte nahoru a dolů, abyste promíchali, nebo použijte špičku prstu, abyste švihli a rovnoměrně promíchali. Dávejte pozor, abyste nemíchali s vírem.

2. Pro konstrukci knihovny musí být použita DNA s vysokou čistotou
■ Dobrá integrita DNA: Pásmo elektroforézy je více než 30 kb, bez ohýbání
■ OD260/230:> 1,5
■ OD260/280: 1,7–1,9
3. Vstupní množství DNA musí být přesné Doporučuje se použít ke kvantifikaci DNA spíše metody Qubit a PicoGreen než Nanodrop.
4. Musí být stanoven obsah EDTA v roztoku DNA. EDTA má velký vliv na fragmentační reakci. Pokud je obsah EDTA vysoký, je nutné před následným testem provést čištění DNA.
5. Fragmentační reakční roztok musí být připraven na ledu. Proces fragmentace je citlivý na reakční teplotu a čas (zejména po přidání zesilovače). Aby byla zajištěna přesnost reakčního času, připravte reakční systém na ledu.
6. Fragmentační reakční čas musí být přesný. Reakční doba fragmentačního kroku bude přímo ovlivňovat velikost fragmentových produktů, čímž ovlivní distribuci velikosti fragmentů DNA v knihovně.
1. Jaký typ vzorku je použitelný pro tuto soupravu?
Použitelným typem vzorku této soupravy může být celková RNA nebo purifikovaná mRNA s dobrou integritou RNA. Pokud je ke konstrukci knihovny použita celková RNA, doporučuje se k odstranění rRNA nejprve použít sadu pro vyčerpání rRNA (kat. Č. 4992363/4992364/4992391).
2. Lze vzorky FFPE použít ke konstrukci knihovny s touto sadou?
MRNA ve vzorcích FFPE bude do určité míry degradována s relativně špatnou integritou. Při použití této sady pro konstrukci knihovny se doporučuje optimalizovat dobu fragmentace (zkrátit dobu fragmentace nebo neprovedení fragmentace).
3. Co by mohlo u kroku výběru velikosti uvedeného v příručce k produktu způsobit mírnou odchylku u vloženého segmentu?
Výběr velikosti musí být prováděn v přísném souladu s krokem výběru velikosti v této příručce k produktu. Pokud dojde k odchylce, důvodem může být to, že magnetické kuličky nejsou vyváženy na pokojovou teplotu nebo nejsou zcela smíchány, pipeta není přesná nebo kapalina zůstává ve špičce. Pro experiment se doporučuje použít tipy s nízkou adsorpcí.
4. Výběr adaptérů při konstrukci knihovny
Stavební sada knihovny neobsahuje činidlo adaptéru a doporučuje se tuto soupravu používat společně s adaptérem TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378).
5. QC knihovny
Kvantitativní detekce knihovny: Qubit a qPCR se používají ke stanovení hmotnostní koncentrace a molární koncentrace knihovny. Operace je striktně v souladu s produktovou příručkou. Koncentrace knihovny bude obecně splňovat požadavky sekvenování NGS. Detekce distribučního rozsahu knihovny: Použití Agilent 2100 Bioanalyzer k detekci distribučního rozsahu knihovny.
6. Výběr čísla amplifikačního cyklu
Podle pokynů je počet cyklů PCR 6-12 a počet potřebných cyklů PCR by měl být vybrán podle vstupu vzorku. V knihovnách s vysokým výtěžkem obvykle dochází k nadměrné amplifikaci v různých stupních, což se projevuje o něco větším píkem po vrcholu cílového rozmezí při detekci Agilent 2100 Bioanalyzer, nebo je detekovaná koncentrace Qubitu nižší než u qPCR. Mírná nadměrná amplifikace je normální jev, který neovlivňuje sekvenování knihovny a následnou analýzu dat.
7. V detekčním profilu bioanalyzátoru Agilent 2100 se objevují hroty
Špičky v detekci bioanalyzátoru Agilent 2100 jsou způsobeny nerovnoměrnou fragmentací vzorků, kde bude v určité velikosti více fragmentů, a to bude zřetelnější po obohacení PCR. V tomto případě se navrhuje neprovádět výběr velikosti, tj. Nastavit podmínky fragmentace na 94 ° C po dobu 15 minut inkubace, kde je distribuce fragmentů malá a koncentrovaná a homogenitu lze zlepšit.
Kategorie produktů
PROČ NÁS VYBRAT
Od svého založení vyvíjí naše továrna prvotřídní výrobky s dodržováním zásad
nejprve kvality. Naše výrobky získaly vynikající pověst v oboru a důvěryhodnost mezi novými i starými zákazníky.